Ідентифікація метилази, необхідної для виробництва 2-метилхопаноїдів, та наслідки для інтерпретації осадових гопанів
Пола В. Веландер
кафедра біології Массачусетського технологічного інституту, 77 Массачусетс-авеню, 68-380, Кембридж, Массачусетс 02139;
Морін Л. Коулман
кафедра біології Массачусетського технологічного інституту, 77 Массачусетс-авеню, 68-380, Кембридж, Массачусетс 02139;
Алекс Л. Сесії
b Відділ геологічних і планетарних наук, Каліфорнійський технологічний інститут, Пасадена, MC100-23, 1200 East California Boulevard, Pasadena, CA 91125;
Роджер Е. Виклик
c Департамент науки про Землю, атмосферу та планети, Массачусетський технологічний інститут, 77 Массачусетс-авеню, E25-633, Кембридж, Массачусетс 02139; і
Діанн К. Ньюман
кафедра біології Массачусетського технологічного інституту, 77 Массачусетс-авеню, 68-380, Кембридж, Массачусетс 02139;
c Департамент науки про Землю, атмосферу та планети, Массачусетський технологічний інститут, 77 Массачусетс-авеню, E25-633, Кембридж, Массачусетс 02139; і
d Медичний інститут Говарда Хьюза, 77 Массачусетс-авеню, 68-171, Кембридж, Массачусетс 02139
Пов’язані дані
Анотація
Результати
Щоб визначити, який ген необхідний для метилювання С-2, ми спочатку ідентифікували передбачувані гени біосинтезу гопаноїдів у геномі TIE-1 R. palustris. В даний час єдиним остаточно відомим етапом біосинтезу гопаноїдів є початкова циклізація сквалену до базової структури хопенового диплоптену сквален-хопенової циклазою (shc). Попереднє дослідження, проведене в Zymomonas mobilis та Bradyrhizobium japonicum, виявило кілька передбачуваних генів біосинтезу гопаноїдів, що оточують локус shc (24). Ми дослідили регіони вище та нижче за течією локусу shc в TIE-1, і виявили подібний передбачуваний кластер біосинтетичних гопаноїдних генів (рис. 1 А). Приблизно на дев'яти кілобазах (kb) вище за геном сквален-ходенциклази ми спостерігали відкриту рамку зчитування (ORF 4269), яка була позначена як передбачуваний B-12-зв'язуючий радикальний білок SAM (рис. 1 A). Оскільки було показано, що клітини R. palustris виробляють 2-метилгопаноїди, мічені в положенні С-2, коли їх годують міченим метіоніном (20, 25), здавалося обґрунтованим, що це метилювання може відбуватися через механізм, залежний від S-аденозилметионіну (SAM). Наявність радикального мотиву SAM у ORF 4269, таким чином, зробило його привабливим кандидатом для метилази.
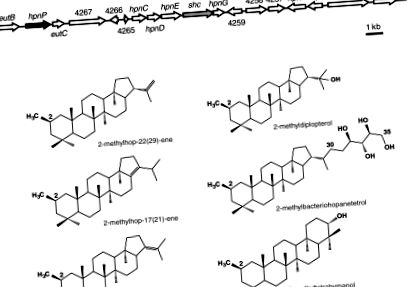
Ідентифікація передбачуваного кластера біосинтетичних гопаноїдних генів у R. palustris TIE-1. (A) Локус shc (Сіра стрілка), необхідний для біосинтезу гопаноїдів, оточений кількома передбачуваними генами біосинтезу гопаноїдів. Вище за SHC був ідентифікований гіпотетичний B-12-зв’язуючий радикальний білок SAM (hpnP, Black Arrow) як кандидат метилази C-2. (B) Структура шести метильованих тритерпеноїдів C-2, вироблених R. palustris TIE-1: три 2-метилхопени (хмель-22 (29) -ен, хмель-17 (21) -ен та хмель-21- ен), 2-метилдиплоптерол, 2-метилтетрагіманол і 2-метилбактеріогопантетрол.
ORF 4269 видаляли в кадрі за допомогою гомологічної рекомбінації, опосередкованої делецією генів, а загальні ліпідні екстракти аналізували за допомогою газової хроматографії-мас-спектрометрії (GC-MS) для отримання метильованих гопаноїдів. Було показано, що R. palustris TIE-1 продукує щонайменше шість різних метильованих тритерпеноїдів С-2 (рис. 1 Б), а отриманий штам делеції не продукував жодної з цих молекул (рис. 2 та рис. S1). Виробництво метильованих гопаноїдів C-2 було відновлено шляхом надання копії гена метилази R. palustris під нативним промотором на самовідтворювальній плазміді (рис. 2). Отже, ORF 4269 - єдиний ген, необхідний для метилювання в положенні С-2, і ми пропонуємо перейменувати локус у hpnP, дотримуючись номенклатури, встановленої в Z. mobilis та B. japonicum (24).
GC-MS загальні іонні хроматограми ацетильованих екстрактів загальних ліпідів культур R. palustris. Мутант ΔhpnP не продукує жодних метильованих гопаноїдів C-2. (A) R. palustris TIE-1. (Б) Р. palustris ΔhpnP. (C) R. palustris ΔhpnP, доповнений геном hpnP на самореплицирующейся плазміді. Пронумеровані сполуки: I, 2-метилгопени; II, надії; III, 2-метилдиплоптерол; IV, диплоптерол; V, 2-метилтетрагиманол; VI, тетрагіманол; VII, 2-метилбактеріогопантетрол; та VIII, бактеріогопантетрол. Ацетильовані загальні ліпідні екстракти аналізували за допомогою високотемпературної ГХ-МС, а сполуки ідентифікували шляхом порівняння часу утримування та мас-спектрів з автентичними сполуками та опублікованих мас-спектрів (таблиця S6). Пік II - це співучасть двох сподіваються структур, hop-22 (29) -ene та hop-21-ene. Жоден метильований гопаноїд не елюювався протягом 29–42 хв (повна хроматограма, показана на рис. S1). Доповнений штам ΔhpnP справді продукував 2-метилбактеріогопантетрол (сполука VII), що підтверджено аналізом масової хроматограми 205 Da та мас-спектру; однак пік занадто широкий, щоб його можна було побачити при роздільній здатності цього показника загального струму іонів GC-MS.
Виникнення генів сквален-ходенциклази (shc) та гепів метилази hpnP у віддалених родинних бактеріях. Дерево представляє філогенетичні взаємозв'язки між бактеріальними видами і було побудовано з максимальною ймовірністю з використанням п'яти об'єднаних основних білків (Adk, RpoB, GyrB, RecA та SecY). Сині смуги у внутрішньому кільці вказують на штами з shc; Червоні смужки у зовнішньому кільці означають штами з hpnP. Наведені всі послідовно виділені ізоляти, що містять shc та/або hpnP, а також представники інших сімейств бактерій. Роди Bacillus (34 ізоляти) та Burkholderia (58 ізолятів) були зруйновані.
Як видно з таблиці S1, організми, що містять метилазу, знаходяться в різних середовищах, включаючи прісну воду, грунт, стічні води, бульбочкові бульбочки та кореневі бульбочки. Відповідно до рідкості 2-метилхопаноїдів у культивованих морських ціанобактеріях (17) жодна із секвенсованих ціанобактерій, що містять ген метилази, не була виділена з морського середовища. Оскільки вважається, що більшість осадових 2-метилгопанів походять із древніх мілководних морських середовищ, де переважають фототрофи (18, 19), ми передбачали, що наш біоінформатичний аналіз може виявити присутність метилази HpnP у ще не перевіреній морській ціанобактерії. Однак α-протеобактерія Nitrobacter sp. Nb-311A був єдиним морським організмом як в геномній, так і в морській метагеномних базах даних, що містив гомолог метилази HpnP.
Щоб зрозуміти походження цього білка, ми реконструювали його філогенез з максимальною вірогідністю та протестували чутливість топології до вирівнювання вхідних даних та вибору таксонів позагрупових груп. Модель заміщення LG (29), обрану ProtTest (30), використовувалась для всіх випробувань, і 10 випадкових стартових дерев використовувались для запобігання пошуку дерев у захопленні в локальному оптимумі. Оскільки якість вирівнювання є головним фактором, що визначає точність дерева (31, 32), ми використовували три програми вирівнювання (CLUSTALW, Muscle та T-Coffee) (33–35). Крім того, ми використали три стратегії фільтрації (розслаблену, сувору та без фільтрації) для усунення невизначених положень вирівнювання (рис. S2). Хоча фільтрація може збільшити відношення сигнал/шум даних, вона також відкидає потенційно інформативні сайти, і це може бути не корисно для коротких вирівнювань (36, 37). Для укорінення дерева ми використовували всіх членів рідної клади радикальних білків SAM як вихідну групу (рис. S3), підхід запропонований як оптимальний (38). Для порівняння в деяких випробуваннях ми додали другу кладу поза групою (рис. S3).
Некореневана топологія групи показує окремі скупчення білка HpnP від видів Methylobacterium, групи Bradyrhizobium/Nitrobacter/Rhodopseudomonas та ціанобактерій (рис. S4). Однак порядок розгалуження цих груп суттєво відрізнявся між випробуваннями. Виникли три різні кореневі топології (рис. 4 та таблиця S3). Найчастіше α-протеобактеріальні послідовності HpnP були монофілетичними і вкладені в лінії послідовностей ціанобактеріальних HpnP, і з цієї топології найбільш ощадливим є висновок про те, що родовий HpnP мешкав у ціанобактеріях (рис. 4 А). У деяких випадках ціанобактеріальні та α-протеобактеріальні послідовності утворювали сестринські плаки (рис. 4 B), або ціанобактеріальні послідовності були вкладені в α-протеобактеріальні послідовності (рис. 4 C). Ця неоднозначність свідчить про те, що філогенетичного сигналу недостатньо для впевненого вирішення порядку розгалуження основних кладів. Ми перевірили, чи найкраща топологія, відновлена для даного вирівнювання, була значно кращою, ніж дві конкуруючі топології, використовуючи тест Приблизно неупереджений (39). Цей тест не зміг відкинути жодної з конкуруючих гіпотез (таблиця S4), і, отже, навіть для даного узгодження ми не можемо впевнено вибрати найкраще дерево.
Філогенетичні взаємозв'язки між метилазами HpnP з трьох віддалених родинних бактеріальних видів. (А) Найкраще дерево, виявлене в дослідженні 15 (таблиця S3), з раннім розгалуженням ціанобактеріального HpnP. (B) Найкраще дерево у дев’ятому дослідженні з монофілетичним ціанобактеріальним HpnP. (C) Найкраще дерево у другому досліді, з раннім розгалуженням Methylobacterium HpnP. Підтримка філій вказується на непараметричну статистику приблизного коефіцієнта правдоподібності (aLRT). Абревіатури роду: M = Methylobacterium, N = Nitrobacter, B = Bradyrhizobium і R = Rhodopseudomonas.
Незалежно від точного порядку розгалуження, наші результати виявляють складну еволюційну історію HpnP. Його філетичний розподіл міг виникнути двома шляхами: або він був присутній у предка ціанобактерій та α-протеобактерій і неодноразово втрачався, або горизонтально переносився між ціанобактеріями та α-протеобактеріями. Переміщення генів між цими тилами спостерігалось (40), і ціанобактерії найчастіше діляться генами з α-протеобактеріями порівняно з іншими тилами (41). У межах ціанобактерій HpnP розподіляється епізодично, а філогенез HpnP не співпадає з філогенезом виду (42). Це передбачає передачу генів та втрату генів серед ціанобактерій, і, отже, неможливо узагальнити, чи всі древні ціанобактерії продукували 2-метилхопаноїди. Позиція самотньої ацидобактерії у дереві HpnP також невизначена. У межах α-протеобактерій HpnP та філогенези видів подібні (43), що передбачає вертикальний спуск у межах цієї групи. З цього філогенетичного аналізу стає ясно, що пошук більшої кількості таксонів для заповнення дерева HpnP є критичним для кращого розв’язання історії цього білка.
Обговорення
У цьому дослідженні ми виявили радикальну SAM-метилазу, кодовану hpnP, необхідну для метилювання С-2 бактеріальних гопаноїдів. В даний час ген hpnP, здається, є надійним предиктором здатності продукувати 2-метилхопаноїди; чи залишатиметься він ексклюзивним провісником, залишається з’ясувати, оскільки в майбутньому буде виявлено більше генів метилази. На сьогодні, однак, використання 2-метилгопаноїдів та осадових 2-метилгопанів як показників ціанобактерій спонукало нас оцінити розподіл продукту гена hpnP у геномах бактерій. Ми виявили, що присутність HpnP не було обмежено цианобактеріями, оскільки це також було виявлено в α-протеобактеріях (зокрема, в ризобіалах) та одній ацидобактерії.
На закінчення, чотири рядки доказів свідчать про те, що 2-метилгопани не можуть бути використані як біомаркери для оксигенного фотосинтезу: i) hpnP розподіляється серед багатьох сучасних бактерій, які не беруть участі в оксигенному фотосинтезі; iii) не всі ціанобактерії утворюють 2-метилхопаноїди (17), і iv) у цианобактерії Nostoc punctiforme, 2-метилхопаноїди локалізуються на зовнішніх мембранах акінетів - структурі виживання, яка не є фотосинтетично активною (23). Вирішення біологічної функції 2-метилгопаноїдів у сучасних бактеріях є необхідним для кращого тлумачення значення 2-метилгопанів у древніх відкладах. Незалежно від їх клітинної функції, застосування (філо) генетики для ідентифікації та інтерпретації історії гена, необхідного для біосинтезу конкретної геостабільної сполуки, демонструє, як два класи молекулярних скам'янілостей - гени та біомаркери - можуть стримувати один одного і забезпечувати більш поінформоване розуміння мікробної еволюції та метаболізму.
Матеріали та методи
Штами бактерій і умови зростання.
Штами бактерій, використані в цьому дослідженні, перелічені в таблиці S5. Штами кишкової палички вирощували в лізогенному бульйоні (LB) при 37 ° C. Штами Rhodopseudomonas palustris вирощували хемогетеротрофно в небуферованому середовищі YP (0,3% дріжджового екстракту, 0,3% пептону) при 30 ° C у темряві при струшуванні при 250 об/хв. Для росту на твердому середовищі LB або YP затвердівали 1,5% агаром і при необхідності додавали гентаміцин при 20 мкг/мл (кишкова паличка) або 800 мкг/мл (R. palustris).
Методи ДНК, побудова плазміди, трансформація та побудова деформацій.
Всі плазмідні конструкції описані в таблиці S5. QIAprep Spin Miniprep Kit (Qiagen) використовували для виділення плазмідної ДНК з кишкової палички. Геномну ДНК із штамів R. palustris виділяли за допомогою набору DNeasy Blood and Tissue Kit (Qiagen). Послідовності ДНК усіх проміжних продуктів, що клонуються, були підтверджені шляхом секвенування в лабораторії біополімерів в Центрі досліджень раку MIT. Штами кишкової палички трансформували шляхом електропорації, використовуючи Electroporator 2510 (Eppendorf), як рекомендував постачальник. Плазміди мобілізували з E. coli S17-1 в R. palustris кон'югацією на агарових пластинах YP, які інкубували протягом ночі при 30 ° C (52, 53). Делеція локусу hpnP в TIE-1 та комплементація мутації делеції виконувались, як описано раніше (15).
Газова хроматографія-мас-спектрометрія (GC-MS).
Для аналізу гопаноїдів штами вирощували до пізньої стаціонарної фази (7 днів). Загальні ліпідні екстракти готували екстракцією розчинником, дериватизували у вигляді ацетатів та аналізували високотемпературною ГХ-МС, використовуючи колонку DB-XLB, як описано раніше (15). З'єднання ідентифікували шляхом порівняння часу утримування та мас-спектрів з автентичними сполуками (тетрагіманол від Trimyema sp .; диплоптерол від Methylococcus capsulatus; бактеріогопанпентол від M. capsulatus) та опубліковані мас-спектри [hopenes: (54); тетрагіманол: (55)]. Метилування С-2 визначали із зсуву фрагмента m/z 191 до 205, m/z 369 до 383 та відносного часу утримування порівняно з дезметильним гомологом (~ 0,15 хв раніше). Часи утримування та молекулярні іони перераховані в таблиці S6.
Філогенез HpnP.
Гомологи HpnP ідентифікували за допомогою BLAST та вирівнювали за допомогою ClustalW, Muscle або T-Coffee (33–35). Вирівнювання були оброблені Gblocks (56) з використанням жорстких (параметри за замовчуванням) або розслаблених налаштувань. У жорсткому випадку не допускалось пропусків у будь-якій послідовності. Для розслабленого випадку мінімальна кількість послідовностей для збережених та флангових позицій була встановлена на мінімально допустимі значення, максимальна кількість неконсервованих позицій була встановлена на 100, мінімальна довжина блоку становила 4, а пропуски допускалися в половині послідовностей. Фіг. S2 показує приклад вирівнювання, сформованого Muscle і обробленого Gblocks. Доцільність двох сімейств позагрупових груп було підтверджено більш вичерпним деревом споріднених білків (рис. S3), що чітко показує, що HpnP та сімейства позагрупових груп є тісно пов'язаними, але різними кладами.
Дерева максимальної вірогідності були побудовані за допомогою PhyML (29), використовуючи гамма-модель LG + [обрана ProtTest (30)], 6 категорій швидкості гамми, 10 випадкових стартових дерев, обмін гілками SPR + NNI та параметри заміщення, оцінені на основі даних. Підтримка філій визначалася за приблизною статистикою LRT, подібною до SH (29). Дерево видів було побудовано аналогічним чином з використанням п'яти об'єднаних білкових послідовностей (RpoB, RecA, Adk, GyrB та SecY) з 375 видів, оброблених за допомогою Gblocks до 1679 позицій та укорінених у Firmicutes. Видове дерево не призначене для вичерпного, але використовується для показу розподілу генів shc та hpnP по бактеріальній філі.
Ми використали Приблизно неупереджений тест (39), реалізований в CONSEL (57), для порівняння топологій дерев. Топології, показані на рис. 4, використовувались як обмеження, а довжини гілок та параметри заміщення були оптимізовані на цих топологіях PhyML. Потім імовірність найкращого необмеженого дерева порівнювали з вірогідністю двох альтернативних дерев, даючи значення р-тесту AU для кожного дерева (таблиця S4). Будь-яке дерево зі значенням р менше 0,05 може бути відхилено. Якщо кілька дерев мають значення p вище цього зрізу, то ми не можемо впевнено вибрати серед них справжнє дерево.
Додатковий матеріал
Подяки.
Ми вдячні Девіду Дауті, Джейкобу Вальдбауеру та Еріку Алму за технічну допомогу та корисні обговорення, а також рецензентів за конструктивні коментарі, які покращили рукопис. Цю роботу підтримали гранти Національної програми з аеронавтики та космічного управління (НАСА) - програма екзобіології (ALS, DKN та RES), Інститут астробіології НАСА (RES), Стипендія Національного наукового фонду після докторських меншин (PVW) та Інститут Агурона Постдокторська стипендія з геобіології (MLC). Д.К.Н. є слідчим медичного інституту Говарда Хьюза.
Виноски
Автори не заявляють конфлікту інтересів.
- Ідентифікація білка з поверхнею LDL, що зв’язує 130 кДа, з клітин гладких м’язів як часткового
- Як налаштувати консула у виробничому середовищі на Ubuntu DigitalOcean
- Для передачі необхідний вільний повнотекстовий Aedes albopictus, пов’язаний з автофагією, ген 8 (AaAtg8)
- Виробництво метаболічного тепла - огляд тем ScienceDirect
- Недостатнє споживання нікотинової кислоти може мати серйозні наслідки для здоров'я